計画研究
![]() 肥後班 「計算科学による核内タンパク質天然変性状態の構造多型解析」
肥後班 「計算科学による核内タンパク質天然変性状態の構造多型解析」
研究代表者:肥後 順一 大阪大学・蛋白質研究所・特任研究員、客員教授
研究分担者:菊池 誠 大阪大学・サイバーメディアセンター・教授
研究分担者:高野 光則 早稲田大学・先進理工学研究科・准教授
研究分担者:菊池 誠 大阪大学・サイバーメディアセンター・教授
研究分担者:高野 光則 早稲田大学・先進理工学研究科・准教授
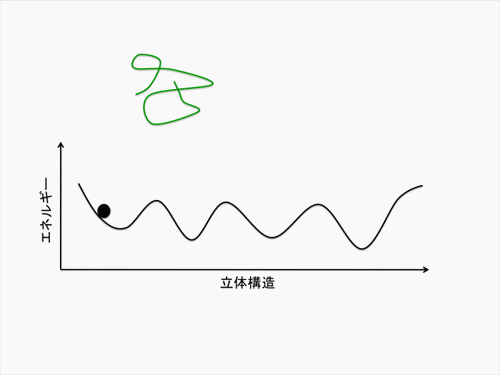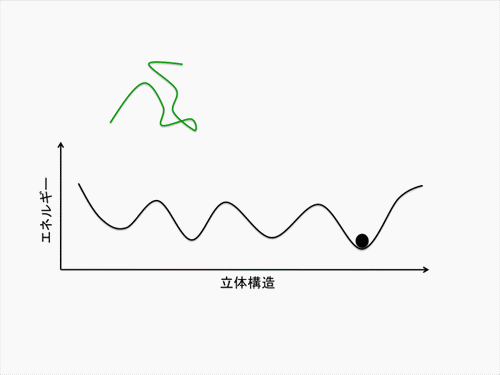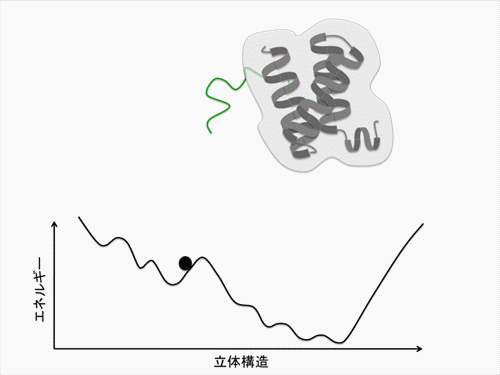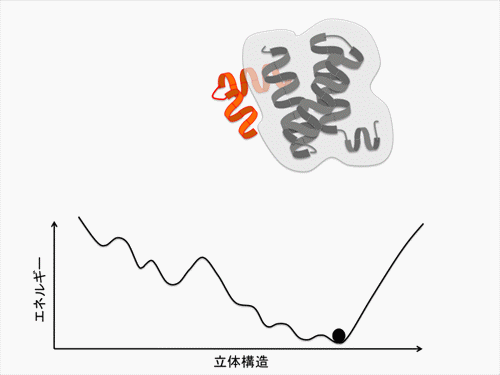
天然変性蛋白質(IDP)は、標的蛋白質への結合と自身の立体構造折れ畳みを不可分なものとして連動させています。IDP単鎖の自由エネルギー地形(自由エネルギーが低いほど、その立体構造は安定)上では、特定の立体構造が安定化されることはありません。しかし、標的蛋白質の存在下では、IDP は複合体を形成するときのみ特定の立体構造を安定化します(アニメ参照)。当計画班は、この結合/折れ畳みの過程をつかさどる自由エネルギー地形を、主に計算機シミュレーションを用いて算出します。
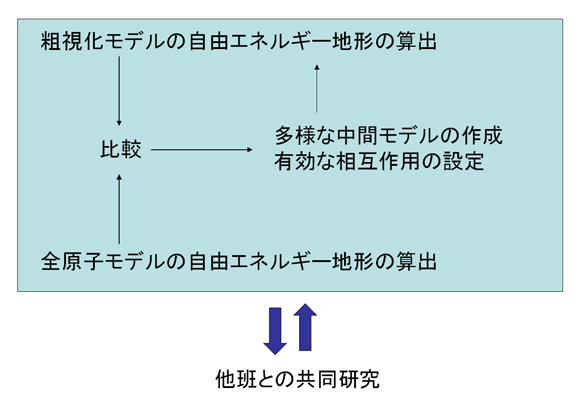
ひとつには水をあらわに取り込んだ全原子モデルを用い、系の多様な立体構造のサンプリングを厳密に行います。あわせて、タンパク質を粗視化したモデルを用いた自由エネルギー地形の算出も行います。さらに粗視化モデルと全原子モデルの間を接続する中間的なモデル(これもある意味粗視化モデルと言えますが)の考案や、原子間(または残基間)相互作用の開発を行い、計算結果をより整合性のあるものにします。
多様な立体構造のサンプリングは、通常のシミュレーションでは実行不可能です。我々はそれをマルチカノニカル法により行います。すなわち、全原子モデルではマルチカノニカル分子動力学法(McMD)を、粗視化モデルではマルチカノニカルモンテカルロ(McMC)法を用います。得られた全ての立体構造を統計力学的に処理することで、室温での自由エネルギー地形を算出します。
以上の手法から得られた計算結果を、実験的な研究結果やデータベースからの予測とすりあわせます。すりあわせを繰り返すことで、IDPの分子認識機構の物理化学的な理解を目指します。